Un nouvel outil d’analyse bio-informatique est en développement dans un laboratoire du Centre hospitalier de l’Université Laval, qui travaille en collaboration avec l’équipe santé du Groupe de recherche en apprentissage automatique de l’Université Laval (GRAAL). Son but ? Rendre accessibles au plus grand nombre possible de chercheurs et chercheuses des analyses biologiques complexes utilisant l’intelligence artificielle.
 Comment soigner un individu si son organisme n’assimile pas correctement un médicament ? Les réactions variables aux traitements sont l’un des problèmes les plus importants dans le domaine de la santé. C’est de là que vient la nécessité de développer de nouvelles méthodes de médecine personnalisée qui permettraient de trouver le bon traitement pour chaque individu. Toutefois, une telle démarche requiert une compréhension extrêmement fine des mécanismes complexes et interreliés du corps humain. Cette compréhension serait bénéfique pour toutes les patientes et les patients, qu’ils soient aux prises avec un cancer, une maladie rare, ou une autre condition. Les traitements pourraient alors être adaptés selon les particularités individuelles, être plus efficaces et sauver davantage de vies.
Comment soigner un individu si son organisme n’assimile pas correctement un médicament ? Les réactions variables aux traitements sont l’un des problèmes les plus importants dans le domaine de la santé. C’est de là que vient la nécessité de développer de nouvelles méthodes de médecine personnalisée qui permettraient de trouver le bon traitement pour chaque individu. Toutefois, une telle démarche requiert une compréhension extrêmement fine des mécanismes complexes et interreliés du corps humain. Cette compréhension serait bénéfique pour toutes les patientes et les patients, qu’ils soient aux prises avec un cancer, une maladie rare, ou une autre condition. Les traitements pourraient alors être adaptés selon les particularités individuelles, être plus efficaces et sauver davantage de vies.
Mon projet vise à développer un algorithme d’intelligence artificielle qui permettra d’atteindre ce niveau de compréhension, puis à le déployer dans une interface Web simple d’utilisation. Sa convivialité rendra cette nouvelle technique accessible à toutes les équipes de recherche des domaines biologiques, et ce, même si elles ne comptent pas de spécialistes en informatique. Ces équipes pourront à leur tour faire avancer leurs recherches et trouver de nouvelles façons de soigner les malades.
Un exemple simple peut illustrer mon projet, celui de la grippe. Un individu ayant le virus de la grippe va probablement tousser, avoir les voies nasales congestionnées ou faire de la fièvre. Ces symptômes sont causés par la réaction du corps à la présence du virus. Or, cette personne ne s’attend pas à être malade (symptômes, durée, etc.) exactement de la même façon que celle qui lui a transmis le virus. Bien que la différence dans les réactions à un stimuli soit considérée comme normale et acceptée, la cause de cette différence, en revanche, est loin d’être simple. L’explication théorique, les scientifiques la connaissent : la façon dont le corps réagit à un événement est principalement guidée par l’ADN, mais elle est aussi modulée par différents « niveaux » biologiques.
Les concepts derrières ces « niveaux » sont complexes. Pour tenter d’y voir plus clair, voyons le corps comme une usine. D’abord, les plans d’un produit sont créés et envoyés vers la chaîne de production. Ensuite, les matières premières sont acquises, les machines, programmées et le personnel, embauché. Enfin, la production commence et des mesures sont mises en place pour soutenir le personnel, par exemple l’apport de bouteilles d’eau et l’ajout de chaises à certains postes. Chaque élément a une fonction bien précise et un problème à n’importe quel point de la chaîne de production pourrait entraîner des effets importants sur l’ensemble.
Revenons à la grippe : ici, l’ADN sert de plans, les métabolites (acquises par l’alimentation) sont les matières premières, les protéines sont les machines et le personnel embauché, et d’autres métabolites sont les bouteilles d’eau et les chaises. Voilà donc les fameux niveaux, qu’on appelle les « omiques », la génomique (l’ADN), la protéomique (les protéines) et la métabolomique (les métabolites).
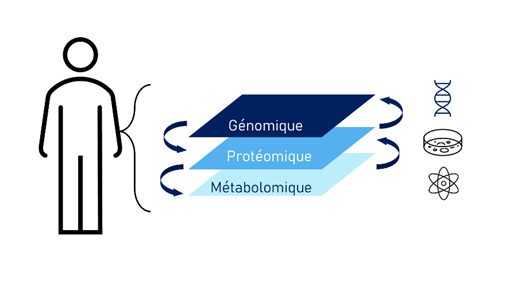
Tout au long de la vie, le corps est exposé à de multiples environnements (la garderie, le milieu de travail, le quartier) qui raffinent les réponses des différentes « omiques ». C’est là que réside toute la complexité du problème : il faut identifier les différences dans chaque niveau et évaluer leurs répercussions sur tous les autres. La clé de l’énigme réside alors dans la compréhension des liens entre les niveaux. Les scientifiques doivent donc décomposer et analyser un enchevêtrement de cascades de réactions.
La métabolomique est un niveau à la fois très intéressant et très difficile à étudier. Elle est surtout pertinente parce qu’elle correspond au niveau le plus proche des symptômes d’une maladie. Les métabolites sont les petites molécules du corps (vitamines, hormones, lipides comme le cholestérol). Ainsi, une mobilisation des ressources de l’organisme, pour se défendre contre un virus tel que la grippe par exemple, a de gros effets, car elle modifie la disponibilité de ces petites molécules ou la façon dont elles circulent. Or, comme on vient de le voir, une modification à un endroit peut entraîner plusieurs répercussions ailleurs. Aussi, la métabolomique est très difficile à étudier parce que les métabolites proviennent de l’environnement et du corps lui-même et non pas d’une source unique. Cependant, en l’intégrant dans une analyse contenant d’autres types de données omiques, on augmente les chances d’avoir une meilleure compréhension de ce qui se passe réellement.
C’est là qu’interviendrait mon interface. Ces liens, ainsi que leurs effets sur les omiques pourront être étudiés en détail, ce qui permettra d’orienter les recherches ou d’ouvrir de nouvelles pistes de solutions.
De plus, cette compréhension de ce qui influence les liens biologiques à l’origine d’une maladie pour un individu donné permettra de rechercher de nouveaux traitements, plus précis, plus adéquats. Mon outil pourra aussi servir de guide pour mieux prévenir certaines maladies, entre autres par le changement de comportements qu’on ne pensait pas dommageables, comme la consommation ou la non-consommation de certains aliments.
Cet article a été réalisé par Élina Francovic-Fontaine, doctorante en médecine moléculaire (Université Laval) avec l’accompagnement de Marie-Paule Primeau, conseillère en vulgarisation scientifique, dans le cadre de notre initiative « Mon projet de recherche en 800 mots ».